Aunque este proceso puede hacerse con la nueva biblioteca terra. Se dice que la biblioteca raster es más eficiente y conveniente para hacer la tarea que nos proponemos resolver, sin embargo, terra está evolucionando y podría ya no ser así. Lo que haremos aquí es leer un montón de archivos raster en formato GeoTiff. Generaremos una colección congruente con ellos en términos de que comparten proyección y extensión y luego haremos una transformación de los objetos de datos para producir una tabla convencional de datos.
Localización de los archivos
En la seción de ayer usamos la biblioteca tools y de ella la función list_files_with_exts. Volvamos a hacerlo.
library("tools")
mapas_dir <- "../../../data/indep_vars"
# Lista de los rasters considerados variables explicativas.
lst_gtif <- list_files_with_exts(mapas_dir, exts = "tif")
lst_gtif [1] "../../../data/indep_vars/AlturaFusteLimpio_desvest_250m.tif"
[2] "../../../data/indep_vars/AlturaFusteLimpio_media_250m.tif"
[3] "../../../data/indep_vars/AlturaTotal_desvest_250m.tif"
[4] "../../../data/indep_vars/AlturaTotal_media_250m.tif"
[5] "../../../data/indep_vars/DiametroCopa_desvest_250m.tif"
[6] "../../../data/indep_vars/DiametroCopa_media_250m.tif"
[7] "../../../data/indep_vars/DiametroNormal_desvest_250m.tif"
[8] "../../../data/indep_vars/DiametroNormal_media_250m.tif"
[9] "../../../data/indep_vars/hemerobia_250m.tif"
[10] "../../../data/indep_vars/net_photo_mean.tif"
[11] "../../../data/indep_vars/net_photo_mean_lluvias.tif"
[12] "../../../data/indep_vars/net_photo_mean_secas.tif"
[13] "../../../data/indep_vars/net_photo_sd.tif"
[14] "../../../data/indep_vars/presenciaArbolesMuertos_250m.tif"
[15] "../../../data/indep_vars/presenciaInsectos_250m.tif"
[16] "../../../data/indep_vars/propor_bosque.tif"
[17] "../../../data/indep_vars/propor_pastizaloagricultura.tif"
[18] "../../../data/indep_vars/propor_selva.tif"
[19] "../../../data/indep_vars/propor_suelodesnudo.tif"
[20] "../../../data/indep_vars/propor_urbano.tif"
[21] "../../../data/indep_vars/srtm90m_mean250m.tif"
[22] "../../../data/indep_vars/srtm90m_range250m.tif"
[23] "../../../data/indep_vars/zvh_31_lcc_h.tif" Pueden ver que hemos encontrado 25 capas de información. Cada una de ellas aportará una columna de datos a nuestra tabla. Para hacer esto lo que haremos es:
- Leer los GeoTiff uno a la vez y agregarlo a una colección brick
- Convertir la colección de objectos raster a una colección de puntos *vectoriales.
- Convertir los puntos en una tabla en la que cada capa aporta una columna.
La tabla que queremos debería tener valores para todas las variablesNoté que rasterToPoints deja pasar algunos datos faltantes que no deberían faltar!!!, Quizás sean resultado de algunas incongruencias en la representación geográfica entre los mapas. Desde luego el tema podría requerir una valoración más cuidadosa, pero para fines prácticos se puede optar por generar una tabla en la que todas las columnas tienen datos. La función de R complete.cases()permite incorporar esta consideración en la construcción de la tabla para omitir todos los registros (casos o renglones) que no tengan valores válidos para todas las variables. Esto lo hace terra en forma predeterminada, así que en este caso también sirve para obtener un resultado comparable.
library(raster)
library(terra)
tiempo_inicio <- Sys.time()
datos_brk <- brick(lst_gtif[1]) # tengo que iniciar el ladrillo, así que lo hago aquí
for (r in lst_gtif[2:length(lst_gtif)]) # como ya use este mapa, el ciclo empieza en el mapa 2
{
datos_brk <- addLayer(datos_brk, raster(r))
}
datos_tbl <- as.data.frame(rasterToPoints(datos_brk)) # aquí ocurre la magia: imágen a tabla!!
datos_tbl <- datos_tbl[complete.cases(datos_tbl), ] # elimina filas con datos incompletos
tiempo_fin = Sys.time()
tiempo_raster <- tiempo_fin - tiempo_inicio
datos_tbl_na <- as.data.frame(rasterToPoints(datos_brk)) # tabla con todo
datos_tbl_na <- datos_tbl_na[!complete.cases(datos_tbl_na),] # tabla con casos incompletosCon fines didácticos y si no por llana curiosidad, les propongo comparar el tiempo que nos toma hacer la tarea que nos propusimos con la biblioteca raster, en contraste con el que nos toma hacerlo con la biblioteca terra.
library(sf)
# La misma tarea de conversión de geotiffs a tabla, pero ahora con terra
tiempo_inicio <- Sys.time()
datos_trr <- rast(lst_gtif) # no necesito un for, ddirectamente procesa la lista de archivos
datos_trr_v <- as.points(datos_trr, na.rm = TRUE) # convierte en vectorial de puntos
datos_trr_sf <- st_as_sf(datos_trr_v, coords = c("x", "y"))
coords_trr <- as.data.frame(st_coordinates(datos_trr_sf$geometry))
datos_trr_df <- as.data.frame(datos_trr_sf)
datos_trr_tbl <- cbind(coords_trr, datos_trr_df)
datos_trr_tbl <- datos_trr_tbl[, -length(datos_trr_tbl)] # elimino la última columna (geometry)
tiempo_fin = Sys.time()
tiempo_terra <- tiempo_fin - tiempo_inicioComparación de tiempos
| Biblioteca | tiempo |
|---|---|
| Raster | 8.67980003356934 |
| Terra | 9.49797582626343 |
Los procesos que aplicamos ¿producen los mismos resultados?. Podemos usar esté código para explorar la cuestión
library(tidyverse)
nombres <- names(datos_trr_tbl) # X, Y van en minúsculas
nombres[1:2] <- c("x", "y")
names(datos_trr_tbl) <- nombres
row.names(datos_trr_tbl) <- NULL # Elimino nombres de fila (dato que no necesito aquí)
row.names(datos_tbl) <- NULL # Elimino nombres de fila (dato que no necesito aquí)
cat("Las tablas raster y terra:\n")Las tablas raster y terra:¿Tienen la misma estructura? TRUE ¿Tienen los mismos datos? TRUE Guardar la tabla en disco
Ahora sólo resta guardar los datos en algun lugar de mi conveniencia. Lo haré en este caso con la biblioteca data.table que es super eficiente para datos grandes, así que conviene que la conozcan. Esta biblioteca contiene la función fwrite que es la que utilizaremos. Crearemos un archivo csv (valores-separados-por-comas). Anote las opciones que requiero para hacer el archivo csv como lo necesito. En R el símbolo “#” indica que lo que sigue la máquina no debe interpretarlo, así que puede usarse para añadir comentarios para el consumo humano.
library(data.table)
fwrite(datos_tbl, # datos obtenidos en el proceso anterior
file = paste0(mapas_dir, "/datos_yuc_2018.csv"), # destino y nombre del archivo
sep = ",", # La coma que separa los valores
quote = FALSE, # No quiero que ponga los nombres entre comillas
na = "*", # Netica utiliza * como indicador de dato faltante
row.names = FALSE, # No quiero una primera columna de números "id"
showProgress = FALSE)# No necesito una barra de progresoCon este proceso hemos preparado una tabla de datos adecuada para ser leída en NETICA y podemos continuar con el procedimiento de construcción de los datos que utilizaremos en la contabilidad.
En Netica se necesitará un pequeño archivo de control que especifica los datos que deseamos recuperar al procesar un nuevo archivo de datos. Para simplificar las cosas lo haremos desde aquí.
Mapear resultados de Netica
Una vez entrenado el modelo de red bayesiana y procesados los casos base para el mapeo, tenemos un mapa de valore predichos. Dada la especificación del archivo de control, ese archivo de salida tendrá una sola columna y tantos renglones como los que tenemos en el archivo de datos utilizaado como fuente de evidencia. El orden de los casos es también el mismo del archivo de datos, así que podemos agregar la nueva comuna del índice de integridad a la tabla que procesamos anteriormente para generar los datos.
ie_dato <- fread(paste0(mapas_dir, "/salida.txt"), header = TRUE, col.names = "iie")
temp <- unique(ie_dato)
# Lo normalizo para expresarlo entre 0 (nula iie) y 100 (alta iie)
ie_dato <- 100 * (18 - ie_dato) / 18
datos_tbl_ie <- cbind(datos_tbl[,c(1:2)], ie_dato)
knitr::kable(head(datos_tbl_ie), caption = "Datos lat-lon e iie")| x | y | iie |
|---|---|---|
| 3935625 | 1135375 | 15.43778 |
| 3932125 | 1135125 | 68.02300 |
| 3932375 | 1135125 | 80.22961 |
| 3932625 | 1135125 | 83.39072 |
| 3932875 | 1135125 | 78.98461 |
| 3933125 | 1135125 | 81.42372 |
Ya tenemos una nueva tabla que incluye coordenadas lat-lon, y el nuevo dato del índice de integridad ecosistémica. Sólo resta convertir estos datos en en una tabla adecuada para representación espacial y a partir de ahí generar el GeoTiff que represente el resultado que obtuvimos.
library (sp)
coordinates(datos_tbl_ie) <- ~ x + y
gridded(datos_tbl_ie) <- TRUE
class(datos_tbl_ie)[1] "SpatialPixelsDataFrame"
attr(,"package")
[1] "sp"plot(datos_tbl_ie)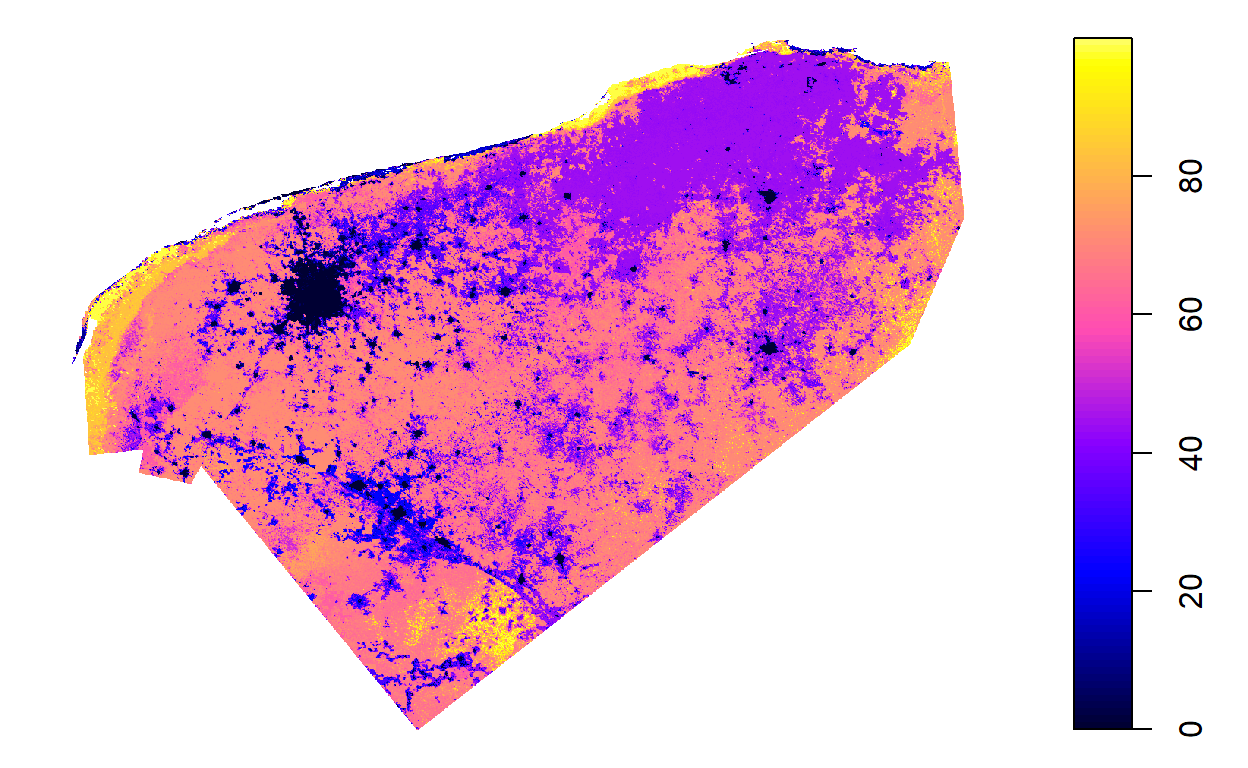
Ahora convierto esta estructura de dato en uun objeto raster, efectivamente será ya el mapa final en memoria. El que guardaremos en disco finalmente.
tamaño x, y del pixel: 250 250writeRaster(datos_ie_r, filename=paste0(mapas_dir, "/../ie/iie_yucatan.tif"),
format="GTiff", overwrite=TRUE)
plot(datos_ie_r)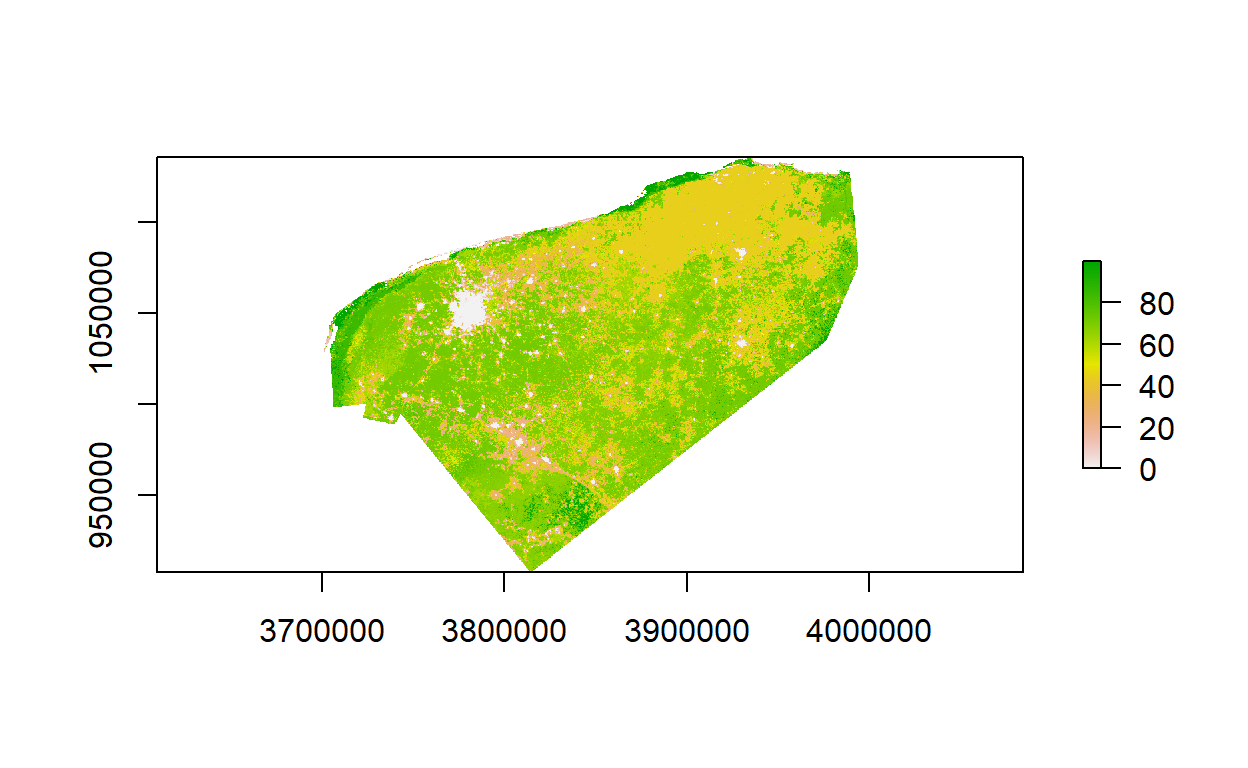
Hemos llegado al final del proceso y ya tenemos el mapa del índice de integridad ecosistémica en el disco.